// Genetische Analysen von potentiellen Lachs- und Forellenhybriden
Zusammenfassung der Masterarbeit von Stefan Theobald (Universität Koblenz-Landau, Campus Landau) unter der Betreuung von Prof. Chris Bridges (TunaTech), und Dr. Anne Schrimpf (Universität Koblenz-Landau), durchgeführt im Rahmen vom Projekt „GeMoLaR“
Dass Kreuzungen zwischen Lachs (Salmo salar) und Bachforelle (S. trutta) möglich sind, ist schon länger bekannt. In den letzten Jahrzehnten wurden verschiedene Methoden entwickelt um Kreuzungen der beiden Arten (Hybride) zu identifizieren. Eine diskutierte Methode ist die Analyse von bestimmten Mikrosatelliten-Marker. Nach einer Theorie von Perrier et al. (2010) sind Hybride daran zu erkennen, dass sie das Allel 162 von der Forelle und das Allel 174 vom Lachs auf dem Mikrosatelliten-Locus D486 aufweisen. Diese Methode ist aber umstritten, da beim Lachs das Allel 162 auch natürlich vorkommen soll. Im Datensatz des GeMoLaR Projektes wurden jedoch 150 Proben identifiziert, die diese beiden Allele tragen. Grund genug, um eine Analyse dieser Proben vorzunehmen und zu schauen, ob oder wie viele Hybride sich in den Daten von GeMoLaR verstecken.
Das erste Ergebnis dieser Arbeit war, dass sich das Allel mit der Länge von 162 Basenpaaren von Lachs und das Allel mit der Länge von 162 Basenpaaren von der Forelle an verschiedenen Stellen durch Mutationen unterscheiden. Wenn eine Probe also das Allel 162 aufweist, ist daher keine Aussage zu treffen, ob es sich bei der Probe um einen ein Hybriden, einen Lachs oder eine Forelle handelt. Abbildung 1 verdeutlicht diese Problematik. Weitere Analysn sind als nötig.
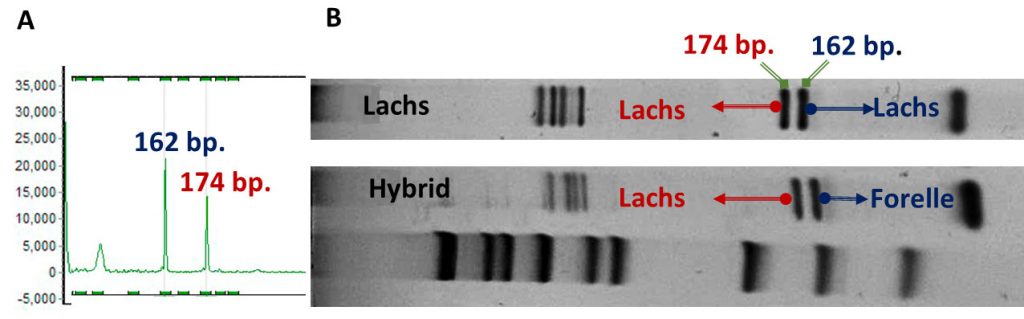
B: Gelelektrophorese zweier Proben deren Banden ausgeschnitten und sequenziert wurden. Die DNA-Sequenzen zeigten,
dass die Banden des Allels 162 im oberen Bild zum Lachs und im unteren Bild zur Forelle gehören, wohingegen das Allel 174
beider Proben dem Lachs zuzuordnen ist. Bei der oberen Probe handelt es sich also um einen Lachs und bei der unteren um einen Hybriden von Lachs und Forelle. Bei der Mikrosatellitenanalyse und auf dem Gel erscheinen die Proben jedoch identisch.
Foto: verändert nach Dr. F. Borutta, 2021.
Im weiteren Verlauf dieser Arbeit wurde zusätzlich der mitochondriale D-Loop, die nuklearen Gene LDH-C und GnRH (Gross et al. 1996) sowie 5S (Pendas et al. 1995) untersucht, um Hybride zu identifizieren. Hierbei wurden die Proben zum Teil sequenziert, aber vor allem mit der Gelelektrophorese untersucht. In Abbildung 2 ist das Gelbild mit zwei Hybriden nach der Methode von Gross et al. (1996) zu erkennen.
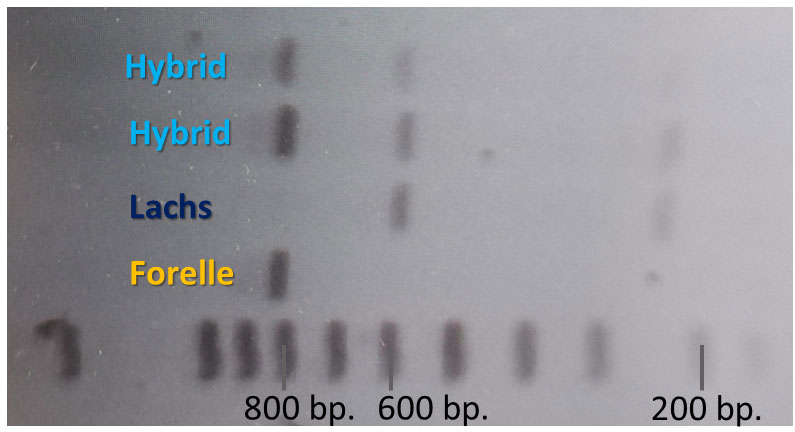
Es ist zu erkennen, dass Hybride die Bandenmuster von Lachs und Forelle aufweisen.
Nach der Analyse von knapp 50 % der Proben wurden drei Hybride, vermutlich der ersten Generation (F1), gefunden. Bei weiteren zwei Proben scheint es sich um post F1 Hybride zu handeln, also um Hybride einer späteren Generation. Es ist festzuhalten, dass weniger Hybride gefunden wurden, als durch die Anzahl der Proben mit dem Allele 162 zu erwarten gewesen wäre. Die Relevanz bleibt aber bestehen, da es sich bei den F1 Hybriden um Tiere aus einer Zucht gehandelt hat und Nachkommen von Hybriden eine geringere Überlebenschance haben.
Die Daten stammen aus folgender Arbeit: Theobald, S. (2022): Genetische Analysen von potenziellen Salmo salar x S. trutta Hybriden. Universität Koblenz-Landau, Institute for Environmental Sciences, Master Thesis, 68p.
Literatur: Perrier, C., et al. (2010). A species-specific microsatellite marker to discriminate European Atlantic salmon, brown trout, and their hybrids. Conservation Genetics Resources 3(1): 131-133.
Gross, R., et al. (1996). A new species-specific nuclear DNA marker for identification of hybrids between Atlantic salmon and brown trout. Journal of Fish Biology 49(3): 537-540.
Pendas, A. M., et al. (1995). Applications of 5S rDNA in Atlantic salmon, brown trout, and in Atlantic salmon x brown trout hybrid identification. Molecular Ecology 4(2): 275-276.
